Introduzione:
L”esercizio fisico, nelle sue varianti, determina molti adattamenti muscolari positivi dal punto di vista morfologico e metabolico, che portano a un miglioramento delle prestazioni e dei tratti legati alla salute. Il muscolo scheletrico è un tessuto plastico in grado di adattarsi rapidamente in risposta a cambiamenti dell’omeostasi metabolica come l’esercizio fisico [1]. Le contrazioni muscolari ripetute determinano un aumento delle dimensioni e del numero dei mitocondri, cambiamenti nel metabolismo dei substrati, una maggiore angiogenesi e l’ipertrofia delle fibre muscolari cardiache e scheletriche [1]. Gli adattamenti all’esercizio fisico avvengono in tempi coordinati e sono mediati da una pletora di regolatori trascrizionali, traslazionali e post-traslazionali [2,3]. Negli ultimi due decenni sono stati compiuti progressi significativi nella ricerca sugli adattamenti cellulari e molecolari all’allenamento muscolare [4]. Tuttavia, nonostante le ricerche in corso, i meccanismi molecolari responsabili di questi adattamenti non sono ancora stati completamente compresi [4,5]. In particolare, non è ben chiaro se e come i segnali epigenetici possano mediare gli adattamenti fisiologici all’allenamento [6,7].
Oltre all’interesse che può portare la conoscenza delle risposte adattative [epigenetica] in risposta all’esercizio fisico, in specie contro-resistenza, il CRISPR gene editing sembra aver portato alcuni a ipotizzare future, e per il momento improbabili applicazioni di questa nuova bio-tecnologia nel BodyBuilding.
Ma prima di proseguire, occorre sapere cosa sia l’epigenetica…
Epigenetica – la potenziale mutazione nel genoma – :
In biologia, l’epigenetica è lo studio dei cambiamenti nell’espressione genica che avvengono senza modifiche alla sequenza del DNA.[Dupont C, Armant DR, Brenner CA (September 2009). “Epigenetics: definition, mechanisms and clinical perspective”] Il prefisso greco epi- (ἐπι- “sopra, al di fuori, intorno”) in epigenetica implica caratteristiche che sono “sopra” o “in aggiunta” al tradizionale meccanismo genetico di eredità (basato sulla sequenza del DNA). [Rutherford A (19 July 2015). “Beware the pseudo gene genies”. The Guardian.] L’epigenetica di solito comporta un cambiamento che non viene cancellato dalla divisione cellulare e influisce sulla regolazione dell’espressione genica.[Deans C, Maggert KA (April 2015). “What do you mean, “epigenetic”?”] Tali effetti sui tratti cellulari e fisiologici possono derivare da fattori ambientali o far parte del normale sviluppo.

In particolare, il termine si riferisce al meccanismo dei cambiamenti: alterazioni funzionalmente rilevanti del genoma che non comportano mutazioni della sequenza nucleotidica. Esempi di meccanismi che producono tali cambiamenti sono la metilazione del DNA e la modificazione degli istoni, ognuno dei quali altera il modo in cui i geni sono espressi senza alterare la sequenza del DNA sottostante.[Kanwal R, Gupta S (April 2012). “Epigenetic modifications in cancer”] Inoltre, è stato dimostrato che le sequenze di RNA non codificanti svolgono un ruolo chiave nella regolazione dell’espressione genica.[Frías-Lasserre D, Villagra CA (2017). “The Importance of ncRNAs as Epigenetic Mechanisms in Phenotypic Variation and Organic Evolution”] L’espressione genica può essere controllata attraverso l’azione di proteine repressori che si attaccano alle regioni silenziatrici del DNA. Questi cambiamenti epigenetici possono durare attraverso le divisioni cellulari per tutta la durata della vita della cellula e possono anche durare per più generazioni, anche se non comportano cambiamenti nella sequenza del DNA sottostante dell’organismo;[Bird A (May 2007). “Perceptions of epigenetics”] invece, fattori non genetici fanno sì che i geni dell’organismo si comportino (o “si esprimano”) in modo diverso.[Hunter P (1 May 2008). “What genes remember”]
Un esempio di cambiamento epigenetico nella biologia eucariotica è il processo di differenziazione cellulare. Durante la morfogenesi, le cellule staminali totipotenti diventano le varie linee cellulari pluripotenti dell’embrione, che a loro volta diventano cellule completamente differenziate. In altre parole, mentre una singola cellula uovo fecondata – lo zigote – continua a dividersi, le cellule figlie risultanti si trasformano in tutti i diversi tipi di cellule di un organismo, compresi i neuroni, le cellule muscolari, l’epitelio, l’endotelio dei vasi sanguigni e così via, attivando alcuni geni e inibendo l’espressione di altri.[Reik W (May 2007). “Stability and flexibility of epigenetic gene regulation in mammalian development”. Nature.]
Questa constatazione rende più complessa la comprensione della trasmissione genetica, introducendo nuovi elementi che rendono sempre meno realistica l’ipotesi dell’evoluzione basati su mutazioni casuali e tassi costanti di cambiamento genetico.
Il cambiamento è intrinseco al genoma. La chiave di “accensione” o “spegnimento” di geni codificanti diversi caratteri è rappresentata dallo stimolo ambientale.
Ma vediamo come questa risposta di attivazione genica si possa manifestare con l’induzione di uno stimolo ambientale specifico come l’allenamento sportivo.
Adattamenti epigenetici all’esercizio fisico:
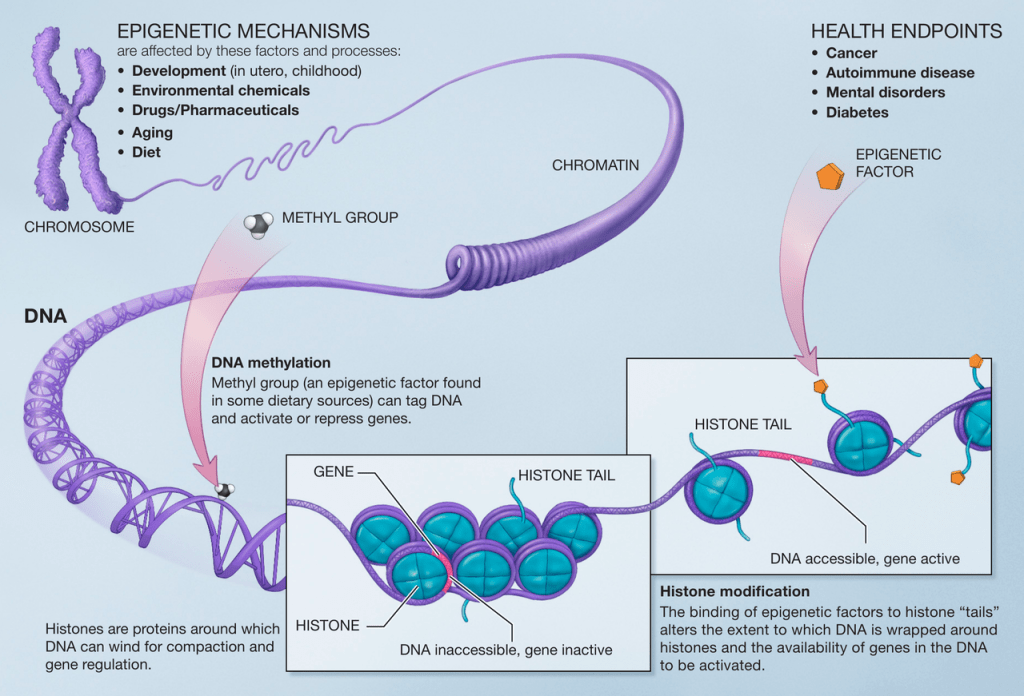
Si ritiene che i meccanismi epigenetici svolgano un ruolo nei complessi adattamenti all’esercizio fisico. L’epigenetica (epi: sopra, genetica: geni) costituisce un importante livello di regolazione dell’espressione genica, senza cambiamenti nella sequenza del DNA. Definiamo una proprietà epigenetica come quella di una cellula, mediata da regolatori genomici, che conferisce alla cellula la capacità di ricordare un evento passato [8]. Le modifiche epigenetiche spesso comportano l’aggiunta di gruppi chimici al DNA o alle code degli istoni e possono essere ereditate attraverso le divisioni cellulari [9]. In questa sede consideriamo due eventi epigenetici principali, la metilazione del DNA e le modifiche degli istoni, insieme a un regolatore epigenetico, gli RNA non codificanti (ncRNA) (ad esempio, i miRNA) . Non consideriamo gli ncRNA come epigenetici di per sé, poiché è difficile capire come i piccoli RNA possano essere ereditabili attraverso la divisione cellulare negli animali [8]. Tuttavia, gli ncRNA possono influenzare l’espressione genica sia a livello trascrizionale che post-trascrizionale senza alterare la sequenza del DNA [10]. Inoltre, il mantenimento dei segni cromatinici è un meccanismo cardine del cambiamento epigenetico e si ritiene che gli ncRNA siano una parte fondamentale di questo processo [11,12]. È interessante notare che alcuni studi hanno dimostrato che le modifiche epigenetiche possono essere modificate in modo tessuto-specifico [9,13,14] da stimoli ambientali come la dieta, il fumo e l’esercizio fisico [5]. È importante notare che alcune modifiche epigenetiche sono transitorie e possono avere una funzione chiave nel muscolo scheletrico.

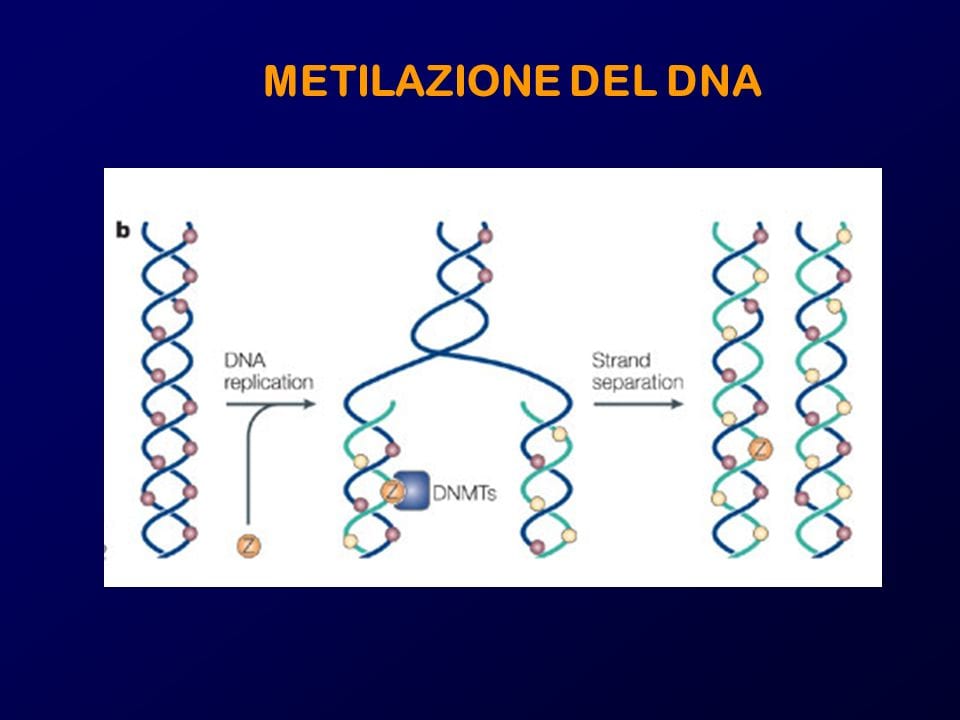
La metilazione del DNA è la modificazione covalente di una base citosinica solitamente situata nella sequenza dinucleotidica 5ʹCpG3ʹ (citosina e guanina separate da un fosfato) [6]. I modelli globali di metilazione del DNA si stabiliscono durante l’embriogenesi nei mammiferi [15] e vengono accuratamente replicati dopo le divisioni cellulari, per cui sono spesso considerati una forma di memoria cellulare [16]. Gli enzimi DNA metiltransferasi (DNMTs), in particolare DNMT3A e DNMT3B, sono responsabili dell’aggiunta del gruppo metile alla base citosina durante la metilazione de novo. L’enzima DNMT1 è poi responsabile del mantenimento dei marchi metilici durante le successive divisioni cellulari [17]. Il fatto che la metilazione del DNA alteri l’espressione genica dipende fortemente dalla posizione genomica all’interno di un gene (cioè promotore, corpo genico o enhancer) e dalla densità delle CpG. Ad esempio, un aumento della metilazione del DNA nei promotori densi di CpG tende a portare a una diminuzione della trascrizione genica [18]. Inoltre, il silenziamento di un gene può portare all’accumulo di metilazione del DNA nel promotore di tale gene, bloccandolo ulteriormente in uno stato di silenziamento [19]. La metilazione del DNA può anche essere rimossa attivamente (demetilazione) dagli enzimi di traslocazione Ten-eleven (TET) [20,21]. Inoltre, esiste un cross-talk tra la metilazione del DNA e altri processi epigenetici, come la metilazione e l’acetilazione della lisina degli istoni [17].
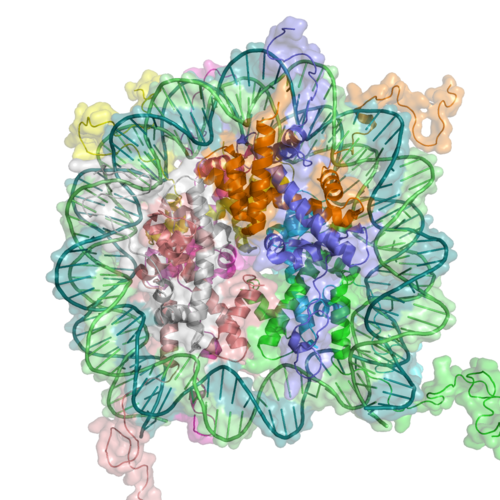
Negli eucarioti, il DNA si avvolge strettamente intorno a proteine chiamate istoni per formare la cromatina [22]. Gli istoni hanno code terminali (N)- e (C)- che sporgono dal centro del nucleosoma e possono interagire con i nucleosomi adiacenti e con il DNA linker [23]. Queste code istoniche possono subire modifiche post-traslazionali (acetilazione, fosforilazione, metilazione e ubiquilazione) che alterano la struttura della cromatina e modificano l’accessibilità dei fattori di trascrizione e dei macchinari al DNA [24]. Le code degli istoni possono anche servire come sito di legame per altre proteine (non istoni) alla cromatina [23]. I geni attivi mostrano tipicamente alti livelli di acetilazione della lisina sulle code degli istoni H3 e H4, trimetilazione delle lisine 4, 36 e 79 di H3 e ubiquitilazione di H2B [23]. Al contrario, i geni che sono stati silenziati mostrano tipicamente la trimetilazione della lisina 9 e 27 di H3 e l’ubiquitilazione della lisina 119 di H2A [23].
L’espressione genica può essere regolata anche da ncRNA [12]. Gli ncRNA meglio caratterizzati sono i microRNA (miRNA), lunghi circa 22 nucleotidi, che mediano il silenziamento genico post-trascrizionale [10]. I miRNA sono molecole non codificanti per le proteine che agiscono tramite accoppiamento di basi alle regioni non tradotte 3 degli mRNA bersaglio e reprimono la sintesi proteica [25]. Circa il 50% dei geni codificanti per le proteine è regolato dai miRNA [26].
I modelli epigenetici sono in parte ereditabili [27], ma sono anche influenzati da fattori ambientali. Uno studio fondamentale condotto su gemelli monozigoti geneticamente identici ha dimostrato che quelli con stili di vita simili presentavano modelli epigenetici simili in più tessuti (linfociti, cellule epiteliali della bocca, grasso intra-addominale e muscolo scheletrico) rispetto ai gemelli monozigoti con stili di vita diversi [28]. L’epigenetica può quindi essere considerata il crocevia tra la genetica (natura) e l’ambiente (cultura) [29]. L’epigenetica è molto promettente per spiegare fenomeni legati all’esercizio fisico, come la variabilità interindividuale a un allenamento simile e la memoria muscolare scheletrica. La variabilità interindividuale si riferisce all’osservazione che, in seguito all’allenamento, alcuni individui migliorano significativamente la loro forma fisica dopo l’intervento (“responders”), mentre altri individui mostrano solo miglioramenti limitati dopo l’intervento (“low-responders”).
L’esercizio fisico è considerato una delle forme di gestione e prevenzione di quasi tutte le malattie croniche più redditizie e sottoutilizzate. L’allenamento all’esercizio fisico può indurre molti cambiamenti molecolari positivi e ridurre la prevalenza di disturbi cognitivi, metabolici, muscoloscheletrici e ossei, oltre ad aumentare il benessere e la salute generale [4]. La programmazione epigenetica può essere modulata dall’esercizio fisico in diversi tessuti, come il muscolo scheletrico [30] e il tessuto adiposo [31]. Dopo un esercizio fisico intenso, le regioni promotrici di geni importanti per il metabolismo dell’esercizio (PGC-1α, PDK4 e PPAR-δ) sono state ipometilate e ciò è stato seguito da un concomitante aumento dei livelli di mRNA. È interessante notare che, 3 ore dopo l’esercizio, questi promotori sono stati ri-metilati, dimostrando la risposta epigenetica dinamica all’esercizio nel muscolo scheletrico [30]. Inoltre, l’espressione di alcuni miRNA cambia dopo l’esercizio, il che potrebbe influenzare la rigenerazione del muscolo scheletrico, la trascrizione genica e la biogenesi mitocondriale [31].
Questa revisione sistematica mirava a identificare i cambiamenti epigenetici segnalati nel muscolo scheletrico a seguito di un esercizio fisico sia acuto (ad esempio una singola sessione di esercizio) che di alcune settimane o mesi di esercizio in popolazioni sane.
Metilazione del DNA ed esercizio fisico – approccio dei geni candidati –
I primi studi sulla metilazione del DNA e l’esercizio fisico hanno analizzato i geni candidati coinvolti nell’adattamento all’esercizio. La maggior parte degli studi sui candidati si è concentrata sul coattivatore 1-alfa del recettore gamma del perossisoma proliferatore attivato (PGC-1a), il regolatore principale della biogenesi mitocondriale e del metabolismo dei grassi [3]. Alibegovic et al. hanno studiato l’effetto del riposo forzato a letto (10 giorni) sui livelli di metilazione del DNA in tre siti CpG situati nel promotore di PGC-1a, nel muscolo vasto laterale di 20 partecipanti [32-42]. La metilazione del DNA in due di questi siti era correlata negativamente con l’espressione dell’mRNA di PGC-1a al basale (sito 816: r = -0,65, P = 0,03; sito 783: r = -0,59, P = 0,04) e la metilazione nel sito 816 è aumentata di circa il 7% dopo il riposo a letto (P = 0,04). Dopo quattro settimane di riallenamento aerobico, i livelli di metilazione del DNA sono diminuiti, ma non sono tornati ai livelli di base [32]. Barres et al. hanno studiato la metilazione del DNA in biopsie del vasto laterale dopo un allenamento acuto e hanno identificato il promotore di PGC-1a come differenzialmente metilato dopo un intervento di esercizio. Inoltre, le regioni del promotore di geni chiave coinvolti nella risposta all’esercizio fisico (PGC-1a, TFAM, MEF2A e PDK4) sono state ipometilate del 10% circa subito dopo un intenso allenamento in bicicletta e sono state rimetilate 3 ore dopo l’esercizio (n = 14, p < 0,05). Va notato che alcuni geni hanno mostrato un’ipometilazione ritardata (ad esempio PPAR-d, 3 ore dopo l’esercizio) e che questi cambiamenti erano dipendenti dall’intensità dell’esercizio. L’ipometilazione dei promotori era accompagnata da aumenti dei livelli di mRNA subito dopo o 3 ore dopo l’esercizio. Bajpeyi et al. hanno suddiviso 11 giovani uomini sani in soggetti ad alta e bassa risposta in base alla risposta di metilazione del DNA in un’importante regione regolatoria di PGC-1a dopo un singolo esercizio fisico [32]. Solo i soggetti high-responder, che presentavano una riduzione della metilazione del DNA dopo l’esercizio, mostravano un riposizionamento dei nucleosomi nel promotore di PGC-1a e un aumento associato dell’espressione dell’mRNA di PGC-1a (da 1,05 ± 0,08 a 1,29 ± 0,11 volte). La dicotomizzazione di soli 11 soggetti inclusi in questo studio è stata arbitraria, il che ha ridotto la potenza statistica e porta a chiedersi se le conclusioni sarebbero state simili utilizzando uno spettro continuo di risposte. Tuttavia, si può concludere che questi studi sui geni candidati hanno dimostrato che l’esercizio fisico altera i livelli di metilazione del DNA nei geni coinvolti nel metabolismo muscolare e sono associati a un cambiamento concomitante nell’espressione dell’mRNA.
Metilazione del DNA ed esercizio fisico – approccio a livello di genoma
Quattro studi hanno condotto studi di associazione epigenomica (Epigenome-Wide Association Studies, EWAS) che hanno analizzato i cambiamenti della metilazione del DNA a livello genomico in seguito a un intervento di allenamento in popolazioni sane [33]. Gli studi consistevano in un intervento di allenamento di resistenza di 6 mesi in 28 uomini di mezza età con e senza storia familiare di diabete di tipo 2 (T2D) [34], in un allenamento di resistenza unilaterale di 3 mesi in 17 giovani uomini e donne [33]; in un intervento di allenamento di resistenza di 7 settimane in 8 giovani uomini [35]; in un allenamento di resistenza, di resistenza o combinato di 3 mesi in 34 uomini e donne giovani e 26 anziani [36]. I risultati di questi studi sono stati molto eterogenei, a causa delle differenze nella modalità di esercizio (resistenza [36], endurance [33,34,36] o entrambi [36]), nella durata dell’intervento (da 7 settimane a 6 mesi), copertura del genoma (solo promotori genici [36] o tutte le regioni genomiche [37-48]), densità di copertura (2-4% di tutte le CpG con gli array Illumina 450 k o 850 k [36] o 8% di tutte le CpG con il MeDIP-chip [34]), dimensione del campione, età e sesso.
Due studi hanno riportato più ipometilazione che ipermetilazione nelle posizioni metilate differenziali (DMP) [34] dopo un intervento di allenamento, uno studio ha riportato un numero simile di DMP ipo- e iper-metilate [33] e l’ultimo studio non ha trovato alcuna DMP [36] dopo 3 mesi di esercizio. È interessante notare che una dimensione moderata dell’effetto è stata coerente tra gli studi (variazione della metilazione < 10% dopo l’intervento [36]), suggerendo che l’allenamento all’esercizio fisico può alterare lo stato di metilazione del DNA di più geni, in modo dipendente dalla dose di esercizio. Sebbene la rilevanza biologica di tali piccoli cambiamenti nella metilazione sia discutibile, è stata dimostrata una correlazione diretta tra i livelli di metilazione del DNA e il conseguente livello di espressione dell’mRNA di geni selezionati mediante un saggio gene reporter [35]. Tre studi hanno identificato una relazione inversa tra la metilazione del DNA e le variazioni dell’espressione genica [33]. Robinson et al., che hanno utilizzato un cut-off assoluto del 5% di variazione della metilazione in seguito all’esercizio fisico, non hanno riscontrato alcuna DMP, ma si sono concentrati solo sulle regioni dei promotori [36]. Lindholm et al. hanno analizzato la distribuzione di queste DMP e hanno riportato un arricchimento di DMP negli enhancer e nelle regioni regolatorie, il che potrebbe spiegare perché non sono state osservate DMP nello studio di Robinson et al. [33]. Vale la pena di notare che l’entità dei cambiamenti nella metilazione del DNA dopo l’allenamento è stata minore rispetto a quella dopo l’esercizio fisico acuto [28], indicando che i cambiamenti nella metilazione del DNA in risposta all’esercizio fisico sono un processo dinamico attivato nella fase iniziale dell’espressione genica. Tuttavia, i cambiamenti residui nella metilazione del DNA si mantengono anche dopo la fine dello stimolo dell’allenamento, indicando che questi cambiamenti si accumulano nel corso di più sessioni di esercizio.
I geni metilati in modo differenziale sono stati arricchiti per vie quali il metabolismo del retinolo e la segnalazione del calcio [34], il rimodellamento strutturale del muscolo, i processi infiammatori/immunologici e la regolazione trascrizionale [33]. Tre degli studi EWAS hanno trovato DMP arricchiti per le vie legate al metabolismo del glucosio e/o dell’insulina [36]. Tuttavia, non è stato studiato a fondo se le modifiche della metilazione del DNA si traducano in cambiamenti a valle del fenotipo. Infatti, solo uno studio ha rilevato che un numero maggiore di siti ipometilati era associato all’ipertrofia muscolare dopo un intervento ripetuto per 7 settimane [36]. Ciò indica che i cambiamenti nella metilazione del DNA potrebbero essere correlati alla capacità di allenamento.
Modificazioni istoniche ed esercizio fisico
Solo uno studio si è concentrato sulle modificazioni degli istoni in seguito all’esercizio fisico. Questo studio ha analizzato i cambiamenti nell’acetilazione di H3K36 e H3K9/14 in nove uomini dopo un allenamento acuto di resistenza [37]. Mentre l’acetilazione di H3K9/14 non è stata alterata, l’acetilazione di H3K36 è aumentata del 64% rispetto al basale (P < 0,05) subito dopo l’esercizio. Poiché l’acetilazione di H3K36 regola l’allungamento trascrizionale, questi risultati suggeriscono che il rimodellamento della cromatina indotto dall’esercizio è associato a un aumento della trascrizione. Mentre non si sono verificati cambiamenti nell’attività globale degli HDAC (P = 0,31), due chinasi che possono indurre l’esportazione nucleare degli HDAC4 e 5 (AMPK e CaMKII) hanno mostrato segni di attivazione. Questi dati delineano una via di segnalazione che potrebbe mediare la trascrizione genica nel muscolo scheletrico umano in risposta all’esercizio fisico [37]. Tuttavia, poiché queste informazioni si basano su uno studio, sono necessarie ulteriori indagini per convalidare questi risultati e scoprire nuove modificazioni istoniche legate all’esercizio.
miRNA e esercizio fisico – approccio dei geni candidati

I miRNA agiscono in modo tessuto-specifico e, quando sono espressi esclusivamente nel muscolo scheletrico, sono chiamati miomiR. Sono stati inclusi in totale 10 studi sui miRNA, suddivisi in studi sui candidati e studi high-throughput. La maggior parte degli studi si è concentrata sui miRNA candidati e sulla loro espressione in seguito all’allenamento, in quanto si tratta di un metodo semplice ed economico per analizzare i miRNA. Sei dei lavori si sono concentrati sull’effetto di un allenamento acuto (resistenza [38], resistenza [39] ed esercizio concomitante [40]), mentre due studi hanno condotto sia un intervento acuto sia un intervento cronico di allenamento [40] (rispettivamente 10 giorni e 12 settimane di allenamento). Inoltre, tre studi hanno analizzato l’effetto dell’allenamento cronico sull’espressione dei miRNA [41] e uno studio ha confrontato atleti di powerlifting con controlli sani [42]. Keller et.al. [42] hanno condotto 6 settimane di allenamento di resistenza, mentre Zhang et.al. e Mueller et.al[41] hanno condotto rispettivamente 20 settimane e 12 settimane di allenamento di resistenza. Sette studi sono stati condotti su uomini, inoltre Zhang et. al. e Mueller et. al. [41] hanno incluso donne nelle loro coorti. Ogni studio aveva una piccola dimensione del campione (8-28 partecipanti), con la più grande coorte combinata di 35 partecipanti sottoposti ad allenamento di resistenza [41]. I cambiamenti nell’espressione dei miRNA dopo l’allenamento dipendono dalla modalità e dalla durata dell’intervento. Dopo l’esercizio acuto, solo il miR-1 e il miR-133a, noti modulatori della proliferazione e della differenziazione muscolare [43], sono stati costantemente upregolati negli studi sui miRNA candidati (p < 0,05) [39]. Tuttavia, dopo l’esercizio fisico cronico, miR-1 e miR-133b sono stati downregolati nella maggior parte degli studi [41]. Solo due studi hanno riportato un aumento dell’espressione del miR-133b e del miR-181, che si pensa sia associato a una maggiore omeostasi del glucosio [44], mentre un altro studio ha riscontrato una diminuzione del miR-23 [38], coinvolto nei processi miogenici [45]. Uno studio caso-controllo che ha confrontato powerlifter e controlli sani [42] ha riportato un profilo di espressione dei miR unico, in grado di distinguere i powerlifter dai controlli sani sulla base di una firma di cinque miR (miR-126, -23b, -16, -23a, -15a). Sebbene siano stati identificati più miRNA associati all’esercizio fisico, i risultati sono stati eterogenei. Le discrepanze tra gli studi potrebbero essere dovute alla variabilità del tempo di biopsia, alla bassa potenza statistica e alle differenze nell’intensità e nella durata dell’esercizio fisico. Inoltre, la quantità variabile di RNA totale può influenzare l’efficienza della sintesi del cDNA [46] e l’uso di diversi geni housekeeping (small RNA a 18s) per la normalizzazione negli studi potrebbe generare risultati variabili [47].
Pochi studi hanno cercato di collegare le variazioni dell’espressione dei miRNA alla capacità di allenamento. Russel et al. [40] hanno riportato correlazioni tra il VO2peak e il Peak Power Output (PPO) con le variazioni dell’espressione dei miRNA. Il VO2peak basale era correlato positivamente con il miR-181 (r = 0,70, P = 0,03), mentre la PPO basale era correlata negativamente con il miR-23a (r = -0,79, P = 0,012) e la PPO post-allenamento era correlata negativamente con il miR-31 (r = -0,74, P = 0,042). Zhang et. al. [36] hanno riportato una forte correlazione positiva tra la variazione della forza del ginocchio in seguito all’allenamento di resistenza e la variazione dell’espressione dei miR-133a, miR-133b e miR-206 (p < 0,01). In sintesi, i cambiamenti di espressione dei miRNA candidati sono stati più consistenti negli studi cronici che in quelli acuti e sembrano dipendere dalla modalità di esercizio, dall’intensità e dalla durata. I cambiamenti nell’espressione dei miRNA in seguito all’allenamento potrebbero essere alla base degli adattamenti all’allenamento, ma sono necessari ulteriori studi per confermarlo.
miRNA e attività fisica – analisi high-throughput
Con i progressi tecnologici, l’analisi dell’espressione dei miRNA ad alto rendimento è diventata più facilmente disponibile con tecnologie che vanno dai microarray, al multiplex digitale e al miRNA-seq, consentendo di analizzare centinaia di miRNA contemporaneamente. Tuttavia, con piattaforme così diverse, i risultati possono produrre differenze di espressione che potrebbero essere semplicemente dovute alla variabilità tra le tecniche. Tutti gli studi high-throughput sono stati condotti dopo un esercizio fisico acuto. Dei quattro studi, tre hanno utilizzato array di miRNA mirati [48] e uno studio ha condotto il sequenziamento di miRNA [49]. Tre degli studi erano basati sull’esercizio di resistenza (uomini, n = 26) [50], e uno dopo un esercizio di resistenza (uomini e donne, n = 6) [48]. La gamma di miRNA differenzialmente espressi nei tre studi variava da 26 a 102 dopo l’esercizio di resistenza [50]. È interessante notare che Zacharewicz et.al. [48] hanno identificato che 7 dei 26 miRNA possono regolare le vie di crescita e proliferazione cellulare e altri nove miRNA possono regolare la via di segnalazione Akt-mTOR, un regolatore centrale della sintesi proteica e della crescita muscolare [51]. McLean et al. [49] hanno trovato 13 miRNA che sono aumentati dopo l’esercizio di resistenza (p < 0,001), e molti di questi miRNA appartengono alla famiglia dei miR-378. Questa famiglia di miRNA è incorporata nel primo introne di PGC-1β [52]. Ogasawara et al. è l’unico studio che ha analizzato i cambiamenti di espressione dei miRNA a livello genomico in seguito all’esercizio fisico cronico e ha scoperto che i livelli di espressione di 102 miRNA erano alterati dopo l’allenamento cronico di resistenza (p < 0,05) [47]. È interessante notare che 26 miRNA sono stati regolati in modo differenziato nei soggetti ad alta e bassa risposta all’ipertrofia [47]. Ciò consolida ulteriormente gli studi sui miRNA candidati, secondo i quali miRNA specifici cambiano in seguito all’esercizio fisico acuto (e forse anche cronico), anche se la funzione specifica dei miRNA nell’allenabilità all’esercizio fisico rimane da chiarire.
Discussione conclusiva sul fattore epigenetico:
Abbiamo in breve riassunto i cambiamenti epigenetici nel muscolo scheletrico in seguito a un allenamento acuto o a un allenamento di poche settimane o pochi mesi in popolazioni sane: metilazione del DNA, miRNA e modifiche degli istoni nel muscolo scheletrico in seguito all’esercizio fisico. Il numero limitato di studi evidenzia quanto sia giovane il campo dell’epigenetica dell’esercizio fisico e che sono necessarie ulteriori indagini su come i segnali epigenetici mediano le risposte all’esercizio.
La programmazione epigenetica può alterare i livelli di espressione genica e migliorare il metabolismo, il che rappresenta uno dei numerosi benefici dell’esercizio fisico per la salute. Tuttavia, la ricerca sulle modifiche epigenetiche nel muscolo scheletrico in seguito all’esercizio fisico è ancora agli inizi e le conseguenze fisiologiche a valle di tali cambiamenti epigenetici devono ancora essere studiate a fondo. Abbiamo esaminato sistematicamente tutti gli articoli pubblicati sull’argomento e avevamo intenzione di condurre una meta-analisi completa a effetti casuali. Tuttavia, a causa dell’esiguo numero di studi e della forte eterogeneità nei disegni degli studi (differenze nelle coorti (sesso, età), nella metodologia (gene candidato, genome-wide), nello stimolo dell’esercizio (acuto/cronico, resistenza/resistenza, alta/bassa intensità, programmi di allenamento lunghi/brevi), non siamo stati in grado di condurre un’analisi quantitativa.
Con lo sviluppo del sequenziamento epigenetico dell’intero genoma e dei metodi analitici, il futuro di questo campo riserva promesse entusiasmanti. Gli studi futuri dovrebbero includere i tessuti chiave influenzati dall’esercizio fisico (ad esempio, muscolo scheletrico e tessuto adiposo), in più punti temporali durante gli interventi di allenamento per costruire un corso temporale delle modifiche epigenetiche durante gli adattamenti all’allenamento e dovrebbero idealmente essere di dimensioni maggiori (n > 50) e di durata maggiore (> 6 mesi). Inoltre, è necessario condurre un lavoro funzionale per dimostrare il coinvolgimento causale delle modifiche epigenetiche indotte dall’esercizio fisico negli adattamenti fisiologici. In sintesi, è stata esaminata sistematicamente la letteratura sulle modifiche epigenetiche nel muscolo scheletrico in seguito all’esercizio fisico e sono stati trovati biomarcatori epigenetici promettenti da validare e replicare in studi futuri. Sono necessarie iniziative multicentriche e collaborative per far progredire il campo e scoprire il significato biologico dei cambiamenti epigenetici nel muscolo scheletrico in risposta all’esercizio.
Adesso passiamo il limite della “genetica imposta” e addentriamoci nelle biotecnologie…
CRISPR gene editing nel Bodybuilding? – la possibile estremizzazione commerciale delle biotecnologie –
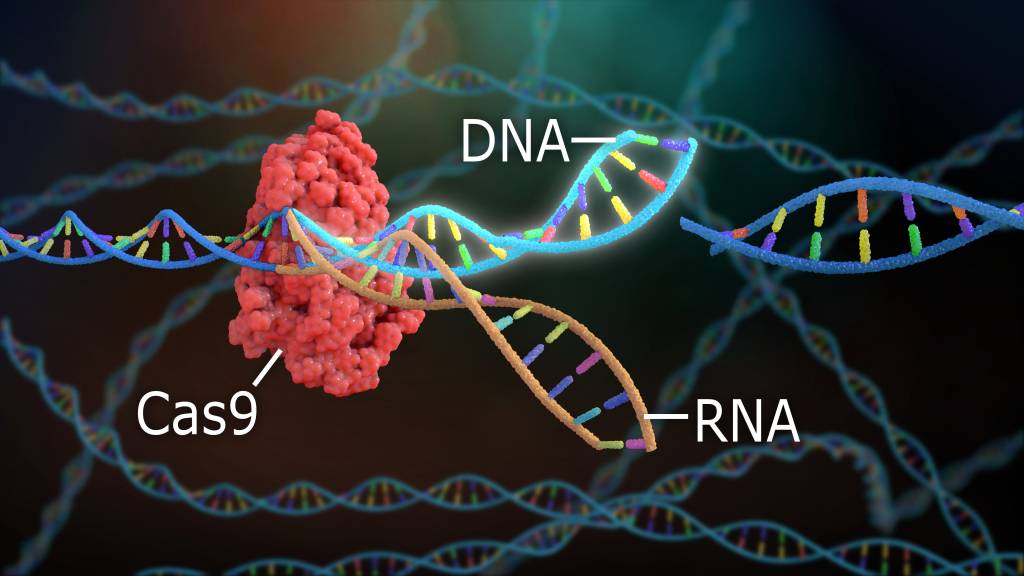
CRISPR gene editing (/ˈkrɪspər/; pronunciato come “crisper”; abbreviazione di “clustered regularly interspaced short palindromic repeats”) è una tecnica di ingegneria genetica in biologia molecolare con cui è possibile modificare i genomi degli organismi viventi. Si basa su una versione semplificata del sistema di difesa antivirale batterico CRISPR-Cas9. Introducendo in una cellula la nucleasi Cas9 complessa con un RNA guida sintetico (gRNA), il genoma della cellula può essere tagliato nel punto desiderato, consentendo di rimuovere i geni esistenti o di aggiungerne di nuovi in vivo.[53]
Questa tecnica è considerata molto importante per la biotecnologia e la medicina, in quanto consente di modificare i genomi in vivo ed è precisa, economica ed efficiente. Può essere utilizzata per la creazione di nuovi farmaci, prodotti agricoli e organismi geneticamente modificati o come mezzo per controllare agenti patogeni e parassiti. Offre inoltre un potenziale nel trattamento di malattie genetiche ereditarie e di malattie derivanti da mutazioni somatiche, come il cancro. Tuttavia, il suo uso nella modificazione genetica della linea germinale umana è molto controverso. Lo sviluppo di questa tecnica è valso a Jennifer Doudna ed Emmanuelle Charpentier il premio Nobel per la chimica nel 2020.[54][55] Il terzo gruppo di ricercatori che ha condiviso il premio Kavli per la stessa scoperta,[56] guidato da Virginijus Šikšnys, non è stato premiato con il Nobel.[57][58][59]

Lavorando come una forbice genetica, la nucleasi Cas9 apre entrambi i filamenti della sequenza mirata di DNA per introdurre la modifica con uno dei due metodi. Le mutazioni Knock-in, facilitate dalla riparazione diretta per omologia (HDR), sono il percorso tradizionale degli approcci di editing genomico mirato.[53] Ciò consente di introdurre un danno al DNA mirato e di ripararlo. L’HDR impiega l’uso di sequenze di DNA simili per guidare la riparazione della rottura attraverso l’incorporazione di DNA esogeno che funge da modello di riparazione.[53] Questo metodo si basa sul verificarsi periodico e isolato di un danno al DNA nel sito bersaglio per avviare la riparazione. Le mutazioni knock-out causate da CRISPR-Cas9 derivano dalla riparazione della rottura a doppio filamento per mezzo della giunzione terminale non omologa (NHEJ) o della POLQ/polimerasi theta-mediated end-joining (TMEJ). Questi percorsi di end-joining possono spesso portare a delezioni o inserzioni casuali nel sito di riparazione, che possono interrompere o alterare la funzionalità del gene. Pertanto, l’ingegneria genomica mediante CRISPR-Cas9 offre ai ricercatori la possibilità di generare un’interruzione genica casuale mirata.
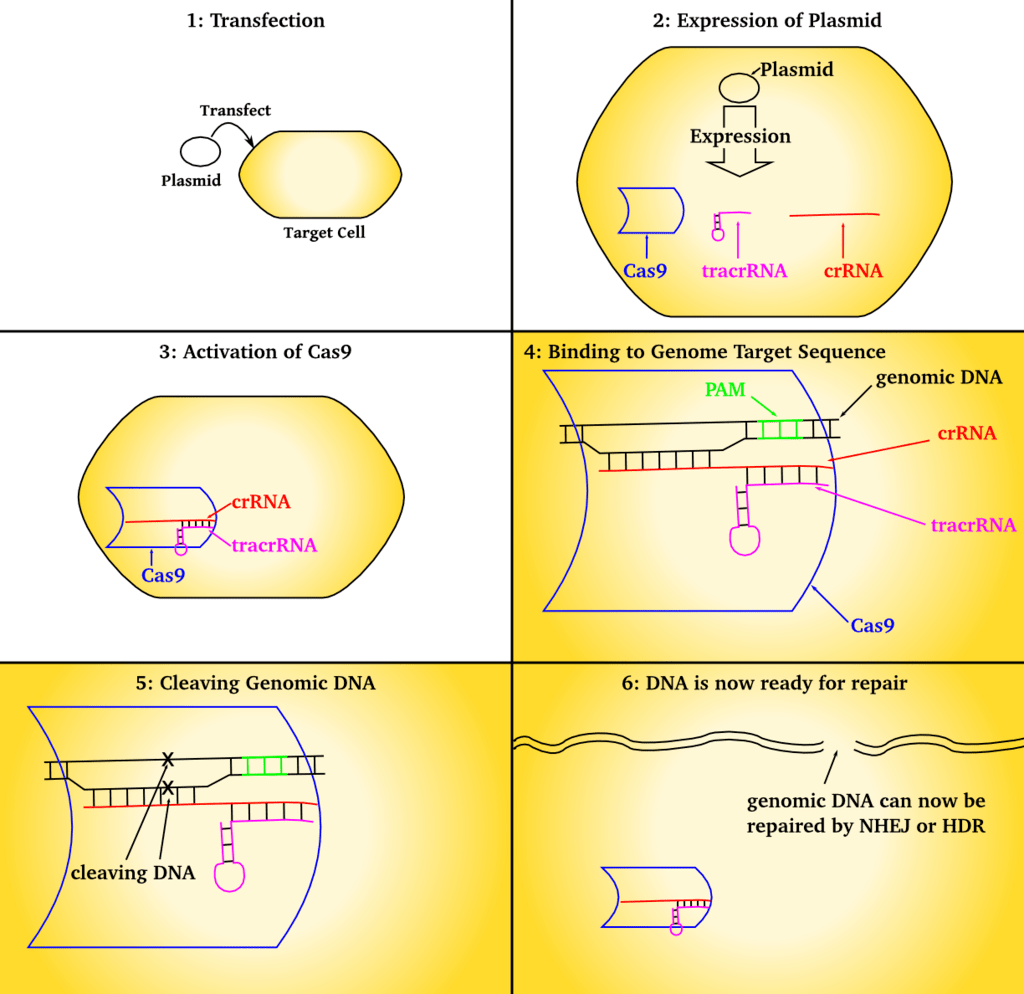
Sebbene l’editing del genoma nelle cellule eucariotiche sia stato possibile con vari metodi fin dagli anni ’80, i metodi impiegati si sono dimostrati inefficienti e poco pratici da implementare su larga scala. Con la scoperta di CRISPR e, in particolare, della molecola di nucleasi Cas9, è diventato possibile un editing efficiente e altamente selettivo. La Cas9, derivata dalla specie batterica Streptococcus pyogenes, ha facilitato la modifica genomica mirata nelle cellule eucariotiche, consentendo un metodo affidabile per creare un’interruzione mirata in un punto specifico, come indicato dai filamenti guida crRNA e tracrRNA.[60] I ricercatori possono inserire la Cas9 e l’RNA modello con facilità per silenziare o causare mutazioni puntiformi in loci specifici. Ciò si è rivelato prezioso per una mappatura rapida ed efficiente di modelli genomici e processi biologici associati a vari geni in una varietà di eucarioti. Sono state sviluppate nuove varianti ingegnerizzate della nucleasi Cas9 che riducono significativamente l’attività fuori bersaglio.[61]

Le tecniche di editing del genoma CRISPR-Cas9 hanno molte applicazioni potenziali. L’uso del complesso CRISPR-Cas9-gRNA per l’editing del genoma[62] è stato scelto dall’AAAS come scoperta dell’anno nel 2015.[63] Sono state sollevate molte preoccupazioni bioetiche sulla prospettiva di utilizzare CRISPR per l’editing della linea germinale, soprattutto negli embrioni umani. [Nel 2023, il primo farmaco che fa uso dell’editing genico CRISPR, Casgevy, è stato approvato per l’uso nel Regno Unito, per curare la malattia falciforme e la beta-talassemia.[64][65] Casgevy è stato approvato per l’uso negli Stati Uniti l’8 dicembre 2023, dalla Food and Drug Administration.[66]

Tutto ciò cosa c’entra con il BodyBuilding? Beh, parlando di una “sottocultura” indirizzata verso l’estremizzazione di determinati caratteri, non era così difficile che alcuni suoi membri pensassero al CRISPR gene editing come ad una futura possibilità di bypassare le limitazioni genetiche individuali.
I “geni del male” che hanno ipotizzato l’uso di questa avanzata biotecnologia per fini estetici, hanno pensato ai seguenti fattori modificabili:
- Sappiamo che la crescita muscolare è in gran parte limitata dal gene della Miostatina, che regola la quantità complessiva di muscoli che un individuo può sintetizzare. Prendendo di mira e riducendo l’attività di questo gene in tutto il corpo, i ricercatori hanno osservato una sostanziale ipertrofia muscolare negli studi sugli animali, con un potenziale simile per gli esseri umani. La modifica dell’espressione della Miostatina potrebbe aprire le porte a dimensioni e densità muscolari senza pari e persino portare a una composizione corporea che resiste naturalmente all’aumento di grasso. Peccato però che ciò si ripercuoterebbe sui sistemi organici e ossei con probabile ipertrofia cardiaca e collasso cardiocircolatorio e/o d’organo;
- Il recupero muscolare e la resistenza alle lesioni sono in parte controllati da geni legati all’infiammazione e alla produzione di collagene. La tecnologia CRISPR potrebbe consentire di modificare questi geni, controllando meglio la risposta infiammatoria dopo allenamenti intensi e rafforzando il tessuto connettivo come tendini e legamenti. Gli atleti potrebbero recuperare dalle sessioni intense con un numero molto inferiore di infortuni, trasformando la loro capacità di allenamento. Ma siamo sicuri di dover proprio attingere ad una biotecnologia come questa per tale ragione? Specie se consideriamo il perfezionarsi delle esecuzioni in linea biomeccanica e la presenza di peptidi specifici per accelerare e migliorare i processi di recupero;
- Geni come PPARγ e UCP1 regolano l’immagazzinamento e l’utilizzo dei grassi. La modifica di questi geni potrebbe facilitare il mantenimento di un bassa body fat senza diete intense o apporti calorici restrittivi, cambiando in modo significativo i processi di cutting e bulking, consentendo ai bodybuilder di aumentare la massa muscolare rimanendo a B.F. basse tutto l’anno. Una condizione o, meglio, una anomalia di questo tipo, porterebbe facilmente ad un alterazione sistemica comprendete anche i meccanismi regolatori della Leptina e della Grelina, tanto per fare un banale esempio. Senza contare le variabili legate alle esigenze organiche in termini di energia, regolazione del metabolismo glucidico in risposta ad un metabolismo lipidico alterato (vedi mutato) ecc…ecc…
- Il recettore degli androgeni (AR), noto anche come NR3C4 (recettore nucleare della sottofamiglia 3, gruppo C, membro 4), è un tipo di recettore nucleare che si attiva legando uno qualsiasi degli ormoni androgeni, tra cui il Testosterone e il DHT, nel citoplasma e traslocando poi nel nucleo. Nell’uomo, il recettore degli androgeni è codificato dal gene AR situato sul cromosoma X a Xq11-12. Da recenti studi, sappiamo che la determinante in quanto a risposta ipertrofica del muscolo scheletrico indotta da AAS è rappresentata dal numero e densità dei AR in misura più incisiva della dose assoluta di AAS. Una modifica in senso iper-espressivo del gene codificante per gli AR sul muscolo scheletrico porterebbe un soggetto ad essere iper-responsivo al trattamento con steroidi anabolizzanti androgeni.
- Il recettore degli estrogeni alfa (ERα), noto anche come NR3A1 (recettore nucleare della sottofamiglia 3, gruppo A, membro 1), è uno dei due tipi principali di recettore degli estrogeni, un recettore nucleare (principalmente come proteina legante la cromatina) che viene attivato dall’ormone sessuale estrogeno. Nell’uomo, ERα è codificato dal gene ESR1 (EStrogen Receptor 1). A differenza del ERβ che può inibire la proliferazione cellulare, l’ERα nel tessuto riproduttivo e mammario ha un azione contraria. L’ERβ è un potente soppressore tumorale e svolge un ruolo cruciale in molti tipi di cancro, come il cancro alla prostata e alle ovaie in contrapposizione all’attività del ERα. Una modifica genica sulla distribuzione dell’espressione del ERα porterebbe il soggetto a tolleranze maggiori di risposta delle alterazioni estrogeniche. Ricordiamoci, però, che l’ERα è altrettanto essenziale nella maturazione e nel mantenimento del fenotipo riproduttivo maschile, poiché i topi maschi ERKO sono sterili e presentano testicoli sottodimensionati. L’integrità delle strutture testicolari dei topi ERKO, come i tubuli seminiferi del testicolo e l’epitelio seminifero, diminuisce nel tempo. Inoltre, le prestazioni riproduttive dei topi maschi ERKO sono ostacolate da anomalie nella fisiologia e nel comportamento sessuale, come l’alterazione della spermatogenesi e la perdita delle risposte intromissorie ed eiaculatorie. Sebbene la sua espressione nell’osso sia moderata, ERα è noto per essere responsabile del mantenimento dell’integrità ossea. Si ipotizza che la stimolazione estrogenica di ERα possa innescare il rilascio di fattori di crescita, come il fattore di crescita epidermico o il fattore di crescita insulino-simile-1, che a loro volta regolano lo sviluppo e il mantenimento dell’osso [oltre ad essere implicato nella regolazione dell’Asse hGH/IGF1]. Nel cervello, ERα si trova nell’ipotalamo, nell’area preottica e nel nucleo arcuato, tutti e tre collegati al comportamento riproduttivo, e la mascolinizzazione del cervello del topo sembra avvenire attraverso la funzione di ERα. Inoltre, studi su modelli di psicopatologia e di malattie neurodegenerative suggeriscono che i recettori estrogenici mediano il ruolo neuroprotettivo degli estrogeni nel cervello.
Inoltre, a proposito di quanto esposto criticamente nei punti sopracitati, sono state elencate alcune sfide pratiche scientifiche delle terapie di editing genico, di cui possiamo evidenziare le due seguenti:
- Interazione genica: Il patrimonio genetico dell’organismo è altamente interconnesso e l’alterazione di un gene potrebbe avere un impatto significativo sugli altri e produrre effetti imprevisti;
- Finalità di applicazione: L’applicazione efficace e mirata della tecnologia di editing genico nell’uomo rimane ancora oggi una sfida significativa. Sebbene l’editing genico sia scientificamente promettente, introduce importanti questioni etiche nello sport agonistico e nella medicina umana. Alterare il DNA per migliorare le prestazioni potrebbe creare un business pronto per essere sfruttato da una delle tante compagnie di biotecnologie con risvolti sociali piuttosto oscuri. Sicuramente, i costi di tale tecnologia la renderebbero accessibile a pochi. Inoltre, i rischi per la salute a lungo termine della modificazione genica di un ente biologico non sono ancora del tutto noti e la modifica del DNA potrebbe portare a effetti collaterali imprevisti, con ripercussioni sulle generazioni future.
Conclusioni sul CRISPR gene editing nel BodyBuilding:
Nel complesso, se dovessimo mettere da parte l’etica e lasciarci andare nel vortice dell’entusiasmo e della speculazione, anche se l’editing genico per il miglioramento delle prestazioni è ancora in fase sperimentale, esso offre aspettative entusiasmanti per il futuro del Bodybuilding e del fitness. Questa tecnologia potrebbe ridefinire l’intera preparazione agonistica, consentendo una nuova era di potenziale fisico oltre i limiti attuali anche per gli atleti enhanced. Se l’editing genico diventerà veramente una possibilità accessibile, il bodybuilding potrebbe evolversi da uno sport dipendente da una disciplina fisica estrema a uno sport profondamente correlato alla biotecnologia, in cui la genetica è progettata per adattarsi al fisico desiderato e agli obiettivi di prestazione dell’atleta. Un passo in avanti verso il così detto “trans-umanesimo”, un passo che se non ben calibrato potrà ritorcersi contro.
Tuttavia, mentre scienziati e filosofi continuano a discutere di queste tecnologie, bisogna riflettere se un ambiente (vedi BodyBuilding e Fitness) già saturo di farmaci per il miglioramento dell’estetica e della performance ha anche solo minimamente bisogno di una biotecnologia dall’enorme potenziale che si trova concretamente giustificata nella sua applicazione solo nei casi di patologie geniche ereditarie o acquisite per mutazione indipendenti.

Dal canto mio, il bisogno non sussiste…
- Interlocutore: Allora è così che si faranno i Bodybuilder.
- Io: No, è così che si gioca a fare Dio. Cioè creare con l’ingegneria genetica dei mostri per dare vita ad una sottospecie umana a tema, né più né meno.
Gabriel Bellizzi [CEO BioGenTech]
Riferimenti:
- [1].Coffey VG, Hawley JA.. The molecular basis of training adaptation. Sport Med. 2007;37:737–763. [DOI] [PubMed] [Google Scholar]
- [2].Hargreaves M. Exercise and gene expression Prog Mol Biol Transl Sci. 2015. p. 457–469. [DOI] [PubMed] [Google Scholar]
- [3].Bishop DJ, Granata C, Eynon N. Can we optimise the exercise training prescription to maximise improvements in mitochondria function and content? Biochim Biophys Acta Gen Subj. 2014;1840:1266–1275. [DOI] [PubMed] [Google Scholar]
- [4].Hawley JA, Hargreaves M, Joyner MJ, et al. Review integrative biology of exercise. Cell. 2014;159:738–749. [DOI] [PubMed] [Google Scholar]
- [5].Soci UPR, Melo SFS, Gomes JLP, et al. Exercise training and epigenetic regulation: multilevel modification and regulation of gene expression Adv Exp Med Biol.. 2017. p. 281–322. [DOI] [PubMed] [Google Scholar]
- [6].Voisin S, Eynon N, Yan X, et al. Exercise training and DNA methylation in humans. Acta Physiol. 2015;213:39–59. [DOI] [PubMed] [Google Scholar]
- [7].Landen S, Voisin S, Craig JM, et al. Genetic and epigenetic sex-specific adaptations to endurance exercise. Epigenetics. 2019; DOI: 10.1080/15592294.2019.1603961. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [8].Lappalainen T, Greally JM. Associating cellular epigenetic models with human phenotypes. Nat Rev Genet. 2017;18:441–451. [DOI] [PubMed] [Google Scholar]
- [9].Weinhold B. Epigenetics: the science of change. Environ Heal Perspect. 2006;114:160–167. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [10].Huang B, Zhang R. Regulatory non-coding RNAs: revolutionizing the RNA world. Mol Biol Rep. 2014;41:3915–3923. [DOI] [PubMed] [Google Scholar]
- [11].Tao H, Yang -J-J, Shi K-H. Non-coding RNAs as direct and indirect modulators of epigenetic mechanism regulation of cardiac fibrosis. Expert Opin Ther Targets. 2015;19:707–716. [DOI] [PubMed] [Google Scholar]
- [12].Peschansky VJ, Wahlestedt C. Non-coding RNAs as direct and indirect modulators of epigenetic regulation. Epigenetics. 2014;9:3–12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [13].Rooney J. Further thoughts on mercury, epigenetics, genetics and amyotrophic lateral sclerosis. 2011;523–524. [DOI] [PubMed]
- [14].van Dijk SJ, Molloy PL, Varinli H, et al. Epigenetics and human obesity. Int J Obes (Lond). 2015;39:85–97. [DOI] [PubMed] [Google Scholar]
- [15].Jaenisch R, Bird A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat Genet. 2003;33:245–254. [DOI] [PubMed] [Google Scholar]
- [16].Sharples AP, Stewart CE, Seaborne RA. Does skeletal muscle have an ’epi’-memory? The role of epigenetics in nutritional programming, metabolic disease, aging and exercise. Aging Cell. 2016;15:603–616. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [17].Li E, Zhang Y. DNA methylation in mammals. Cold Spring Harb Perspect Biol. 2014;1;6:a019133. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [18].Nan X, Ng HH, Johnson CA, et al. Transcriptional repression by the methyl-CpG-binding protein MeCP2 involves a histone deacetylase complex. Nature. 1998;393:386–389. [DOI] [PubMed] [Google Scholar]
- [19].Ohm JE, McGarvey KM, Yu X, et al. A stem cell-like chromatin pattern may predispose tumor suppressor genes to DNA hypermethylation and heritable silencing. Nat Genet. 2007;39:237–242. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [20].Jones PA. Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat Rev Genet. 2012;13:484–492. [DOI] [PubMed] [Google Scholar]
- [21].Guibert S, Weber M. Functions of DNA methylation and hydroxymethylation in mammalian development. Curr Top Dev Biol. 2013;104:47–83. [DOI] [PubMed] [Google Scholar]
- [22].Bentley GA, Lewit-Bentley A, Finch JT, et al. Crystal structure of the nucleosome core particle at 16A resolution. J Mol Biol. 1984;176:55–75. [DOI] [PubMed] [Google Scholar]
- [23].Zhang T, Cooper S, Brockdorff N. The interplay of histone modifications – writers that read. EMBO Rep. 2015;16:1467–1481. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [24].Bannister AJ, Kouzarides T. Regulation of chromatin by histone modifications. Cell Res. 2011;21:381–395. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [25].Hussain MU. Micro-RNAs (miRNAs): genomic organisation, biogenesis and mode of action. Cell Tissue Res. 2012;349:405–413. [DOI] [PubMed] [Google Scholar]
- [26].Widmann M, Nieß AM, Munz B. Physical exercise and epigenetic modifications in skeletal muscle. Sport Med. 2019. [DOI] [PubMed] [Google Scholar]
- [27].McClay JL, Shabalin AA, Dozmorov MG, et al. High density methylation QTL analysis in human blood via next-generation sequencing of the methylated genomic DNA fraction. Genome Biol. 2015;16:291. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [28].Fraga MF, Ballestar E, Paz MF, et al. Epigenetic differences arise during the lifetime of monozygotic twins. Proc Natl Acad Sci U S A. 2005;102:10–15. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [29].van Dongen J, Nivard MG, Willemsen G, et al. Genetic and environmental influences interact with age and sex in shaping the human methylome. Nat Commun. 2016;7:11115. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [30].Barrès R, Yan J, Egan B, et al. Acute exercise remodels promoter methylation in human skeletal muscle. Cell Metab. 2012;15:405–411. [DOI] [PubMed] [Google Scholar]
- [31].Fabre O, Ingerslev LR, Garde C, et al. Exercise training alters the genomic response to acute exercise in human adipose tissue. Epigenomics. 2018;10:1033–1050. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [32].Russell AP, Lamon S, Boon H, et al. Regulation of miRNAs in human skeletal muscle following acute endurance exercise and short-term endurance training. J Physiol. 2013;591:4637–4653. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [33].Moher D, Shamseer L, Clarke M, et al. Preferred reporting items for systematic review and meta-analysis protocols (PRISMA-P) 2015 statement. Syst Rev. 2015;4:1–19. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [34].Alibegovic AC, Sonne MP, Højbjerre L, et al. Insulin resistance induced by physical inactivity is associated with multiple transcriptional changes in skeletal muscle in young men. Am J Physiol – Endocrinol Metab. 2010;299:752–763. [DOI] [PubMed] [Google Scholar]
- [35].Bajpeyi S, Covington JD, Taylor EM, et al. Skeletal muscle PGC1alpha −1 nucleosome position and −260nt DNA methylation determine exercise response and prevent ectopic lipid accumulation in men. Endocrinology. 2017;158:2190–2199. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [36].Lindholm ME, Marabita F, Gomez-Cabrero D, et al. An integrative analysis reveals coordinated reprogramming of the epigenome and the transcriptome in human skeletal muscle after training. Epigenetics. 2014;9:1557–1569. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [37].Nitert MD, Dayeh T, Volkov P, et al. Impact of an exercise intervention on DNA methylation in skeletal muscle from first-degree relatives of patients with type 2 diabetes. Diabetes. 2012;61:3322–3332. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [38].Seaborne RA, Strauss J, Cocks M, et al. Human skeletal muscle possesses an epigenetic memory of hypertrophy. Sci Rep. 2018;8:1898. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [39].Robinson MM, Dasari S, Konopka AR, et al. Enhanced protein translation underlies improved metabolic and physical adaptations to different exercise training modes in young and old humans. Cell Metab. 2017;25:581–592. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [40].Lindholm ME, Giacomello S, Werne Solnestam B, et al. The impact of endurance training on human skeletal muscle memory, global isoform expression and novel transcripts. PLOS Genet. 2016;12:e1006294. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [41].McGee SL, Fairlie E, Garnham AP, et al. Exercise-induced histone modifications in human skeletal muscle. J Physiol. 2009;587:5951–5958. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [42].Nielsen S, Scheele C, Yfanti C, et al. Muscle specific microRNAs are regulated by endurance exercise in human skeletal muscle. J Physiol. 2010;588:4029–4037. [DOI] [PMC free article] [PubMed] [Google Scholar
- 43.Ringholm S, Biensø RS, Kiilerich K, et al. Bed rest reduces metabolic protein content and abolishes exercise-induced mRNA responses in human skeletal muscle. Am J Physiol Endocrinol Metab. 2011;301:E649–58. [DOI] [PubMed] [Google Scholar]
- [44].Rivas DA, Lessard SJ, Rice NP, et al. Diminished skeletal muscle microRNA expression with aging is associated with attenuated muscle plasticity and inhibition of IGF-1 signaling. Faseb J. 2014;28:4133–4147. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [45].D’Souza RF, Markworth JF, Aasen KMM, et al. Acute resistance exercise modulates microRNA expression profiles: combined tissue and circulatory targeted analyses. PLoS One. 2017;12e0181594. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [46].Fyfe JJ, Bishop DJ, Zacharewicz E, et al. Concurrent exercise incorporating high-intensity interval or continuous training modulates mTORC1 signaling and microRNA expression in human skeletal muscle. Am J Physiol – Regul Integr Comp Physiol. 2016;310:R1297–311. [DOI] [PubMed] [Google Scholar]
- [47].Keller P, Vollaard NBJ, Gustafsson T, et al. A transcriptional map of the impact of endurance exercise training on skeletal muscle phenotype. J Appl Physiol. 2011;110:46–59. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [48].Zhang T, Birbrair A, Wang ZM, et al. Improved knee extensor strength with resistance training associates with muscle specific miRNAs in older adults. Exp Gerontol. 2015;62:7–13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [49].Mueller M, Breil FA, Lurman G, et al. Different molecular and structural adaptations with eccentric and conventional strength training in elderly men and women. Gerontology. 2011;57:528–538. [DOI] [PubMed] [Google Scholar]
- [50].D’Souza RF, Bjørnsen T, Zeng N, et al. MicroRNAs in muscle: characterizing the powerlifter phenotype. Front Physiol. 2017;8:1–12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [51].Chen J-F, Mandel EM, Thomson JM, et al. The role of microRNA-1 and microRNA-133 in skeletal muscle proliferation and differentiation. Nat Genet. 2006;38:228–233. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [52].Sun X, Lin J, Zhang Y, et al. MicroRNA-181b improves glucose homeostasis and insulin sensitivity by regulating endothelial function in white adipose tissue. Circ Res. 2016;118:810–821. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 53 Bak RO, Gomez-Ospina N, Porteus MH (August 2018). “Gene Editing on Center Stage”. Trends in Genetics. 34 (8): 600–611. doi:10.1016/j.tig.2018.05.004. PMID 29908711. S2CID 49269023.
- 54 “The Nobel Prize in Chemistry 2020”. The Nobel Prize. Retrieved 2020-12-10.
- 55 Cohen J (October 7, 2020). “CRISPR, the revolutionary genetic “scissors,” honored by Chemistry Nobel”. Science. doi:10.1126/science.abf0540. S2CID 225116732.
- 56 Cohen J (2018-06-04). “With prestigious prize, an overshadowed CRISPR researcher wins the spotlight”. Science | AAAS. Retrieved 2020-05-02.
- 57 Jump up to:a b Owens R (8 October 2020). “Nobel prize: who gets left out?”. The Conversation. Retrieved 13 December 2021.
- 58 “Lithuanian scientists not awarded Nobel prize despite discovering same technology”. LRT.LT. 8 October 2020.
- 59 Šikšnys V (2018-06-16). “Imam genų žirkles, iškerpam klaidą, ligos nelieka”. Laisvės TV / Freedom TV. 12:22 minutes in. LaisvėsTV. <…>Tai mes tą savo straipsnį išsiuntėm į redakciją pirmieji, bet laimės ten daug nebuvo. Viena redakcija pasakė, kad mes net recenzentam nesiųsim. Nusiuntėm į kitą redakciją – tai jis (straipsnis) pragulėjo kažkur ant redaktoriaus stalo labai ilgai. Na ir taip galų gale išsiuntėm į trečią žurnalą ir trečias žurnalas po kelių mėnesių jį išspausdino. Bet, aišku, Berklio universiteto mokslininkams sekėsi geriau – jie išsiuntė straipsnį į žurnalą Science – jį priėmė ir išspausdino per 2 savaites. Nors iš tikro jie tą straispnį išsiuntė pora mėnesių vėliau nei mes. Retrieved 2018-06-30.
<…> Well, we were who had sent the article first, but had not much of luck.
- 60 Zhang JH, Pandey M, Kahler JF, Loshakov A, Harris B, Dagur PK, et al. (November 2014). “Improving the specificity and efficacy of CRISPR/CAS9 and gRNA through target specific DNA reporter”. Journal of Biotechnology. 189: 1–8. doi:10.1016/j.jbiotec.2014.08.033. PMC 4252756. PMID 25193712.
- 61 Vakulskas CA, Dever DP, Rettig GR, Turk R, Jacobi AM, Collingwood MA, et al. (August 2018). “A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells”. Nature Medicine. 24 (8): 1216–1224. doi:10.1038/s41591-018-0137-0. PMC 6107069. PMID 30082871.
- 62 Jump up to:a b c Ledford H (March 2016). “CRISPR: gene editing is just the beginning”. Nature. 531 (7593): 156–159. Bibcode:2016Natur.531..156L. doi:10.1038/531156a. PMID 26961639.
- 63 Travis J (17 December 2015). “Breakthrough of the Year: CRISPR makes the cut”. Science Magazine. American Association for the Advancement of Science.
- 64 Ledford H (June 2015). “CRISPR, the disruptor”. Nature. 522 (7554): 20–24. Bibcode:2015Natur.522…20L. doi:10.1038/522020a. PMID 26040877.
- 65 Jump up to:a b “Casgevy: UK approves gene-editing drug for sickle cell”. BBC News. 16 November 2023. Retrieved 16 November 2023.
- 66 Jump up to:a b “MHRA authorises world-first gene therapy that aims to cure sickle-cell disease and transfusion-dependent β-thalassemia”. Gov.uk. 16 November 2023. Retrieved 16 November 2023.
